摘 要 Abstract
现代生物技术的快速发展使生物医药行业发生了革命性的变化。疫苗的开发虽已取得了诸多成果,但仍有很多疾病不能预防,新兴技术加快了疫苗的研发速度,有望成为开发针对这些疾病疫苗的有效手段。随着全基因组测序的迭代更新和生物信息学的兴起,反向疫苗学技术应运而生。该技术以基因组序列为目标,预测疫苗的所有候选抗原。本文主要对反向疫苗学技术的背景以及反向疫苗学技术“1.0时代”和“2.0 时代”技术路线进行概述。
The rapid development of modern biotechnology has revolutionized the biomedical industry. Although many achievements have been made in the development of vaccines, there are still many diseases that cannot be prevented.Emerging technologies have accelerated the development of vaccines, thus promising to be an effective means of developing vaccines against these diseases. With the iterative update of whole genome sequencing and the rise of bioinformatics, reverse vaccinology technologies have emerged. This technique targets genomic sequences to predict all candidate antigens for vaccines. This article mainly summarizes the background of reverse vaccinology technology and the technical route of “1.0 era” and “2.0 era” of reverse vaccinology technology.
1、 前言
反向疫苗学(Reverse Vaccinology)是从病原体基因组序列出发,应用生物信息学技术、蛋白质组学技术、生物芯片技术等工具,预测病原体的毒力因子、外膜抗原、侵袭及毒力相关抗原;应用抗原表位预测工具筛选出能够引起免疫应答的抗原表位分子,对上述抗原基因进行高通量克隆、表达、纯化出重组蛋白,然后再对纯化后的抗原进行体内、体外评价,筛选出保护性抗原进行疫苗研究,这种以基因组为基础,筛选蛋白质抗原的疫苗研发策略称为反向疫苗学技术[1-3]。
1.1 背景
疫苗是控制及消灭病原体的一种有效手段。传统疫苗包括灭活疫苗、减毒活疫苗、类毒素疫苗和亚单位疫苗。20 世纪应用传统技术研制的疫苗相继问世,对许多重要疾病的有效防治起到了关键性作用,如脊髓灰质炎、伤寒、麻疹、腮腺炎、乙型脑炎等疾病。但是对于研究难度较高的复杂病原体,很难通过传统的方法制备高效安全的疫苗,如某些病原体无法在体外培养且易变异、抗原体外表达量小不足以分离纯化或免疫原性微弱等,即便利用传统方法可以制备,仍面临提纯工艺复杂、成本高等困境,以致目前仍有不少严重的感染性疾病尚无疫苗可用[4]。
基因组携带着生物的遗传信息,通过测序技术可以确定基因组内的碱基排列顺序, 而测序技术的迅猛发展也使得通过病原体全基因组信息来找寻新的抗原变为现实。1953 年, 沃森(Watson)和克里克(Crick)提出DNA 双螺旋结构,为测序技术的出现奠定了基础。1977 年,桑格(Sanger)发明双脱氧测序法,这是第一代测序技术。1995年, 温特(Venter) 等人完成了流感嗜血杆菌(haemophilus influenzae)的全基因组测序[5],2000 年他们对B 群脑膜炎奈瑟菌(Neisseria meningitidis group B)MC58 株的全部基因组进行了注释,从中发现了大量新的表面抗原[6-7]。这种从病原体全基因组信息出发进行疫苗研究的新策略被称之为“反向疫苗学”,如图1 所示。它能帮助研究人员克服常规疫苗方法的限制,这在多种病原体疫苗研发中得以体现,其中B 群脑膜炎奈瑟菌疫苗即为首个利用反向疫苗学技术研究的成功案例[8]。

在复杂病原体的疫苗研发过程中,仍有一些技术难点无法攻克。2002 年,伯顿(Burton)等人提出了一种通过抗体信息设计新抗原的方法,即分离患者体内的特异性B 细胞,体外建立抗体组库,筛选有效的中和抗体,通过抗原抗体复合物的结构解析,找到关键性抗原表位,最终设计出具有高保护效果的疫苗[9]。2008年,拉波利(Rappuoli)等人提出了一种基于结构信息优化抗原设计的新思路,通过对抗原结构进行分析,设计出稳定性好、产量高及免疫原性优的抗原[10]。呼吸道合胞体病毒(respiratorysyncytial virus,RSV) 的F 蛋白抗原设计就是比较经典的例子。F 蛋白有两种构象——融合前构象(Pre-F)和融合后构象(Post-F)。虽然Pre-F 的稳定性不如Post-F,但是其能够诱导更高水平的中和抗体。麦克莱伦(McLellan)等人通过改造F 蛋白跨膜区获得一个稳定的三聚体Pre-F 蛋白,该蛋白能够诱导更高水平的中和抗体[11],为宿主提供更全面的保护。得益于蛋白质组学和结构生物学的发展,反向疫苗学技术从“1.0 时代”跨入“2.0时代”[12-13]。
1.2 优势与局限性
传统疫苗学是建立在充分认识病原体生物学性状、致病性以及免疫原性等特性的基础上,并且需要在体外进行大规模培养。对于一些具有严重传染性和致病性的病原体来说,体外培养具有极大的安全隐患,这些因素使得传统疫苗学方法存在较大的局限性。而反向疫苗学技术从病原体的基因组序列入手,与传统的疫苗研制思路截然不同,具有以下优势:①反向疫苗学技术从分析基因组序列开始,就不需要在体外进行病原体的培养,避免了病原体的扩散;②反向疫苗学技术对病原体的所有蛋白质都进行了分析预测,增加了筛选范围,能筛选到部分被忽略的候选抗原;③反向疫苗学技术可直接对病原体的疫苗靶点进行预测,极大地缩减了早期研发的时间和成本。
当然,目前反向疫苗学技术还存在一定的局限性。例如,抗原预测种类比较单一,大多数是蛋白质分子。而在传统疫苗学的研究中发现,有许多非蛋白质分子,如多糖、脂类等也具有良好的免疫原性,同时还能起到类似佐剂的效果。而这部分分子尚无法通过反向疫苗学技术进行筛选。其次,基因组学和生物信息学中所使用的算法准确度仍存在一定的局限性,需要进一步优化,以提高有效抗原预测的准确性。另外,反向疫苗学技术应用通用表达系统表达的候选疫苗分子与病原体体内天然蛋白分子的构象和修饰存在一定差别,因此,它们的免疫原性和保护力存在差异,可能会影响研究人员的判断和抗原筛选的准确度。
2、 候选抗原的筛选与预测
在传统疫苗学的研究和开发过程中发现,病原体表面的分子和病原体分泌的蛋白质能够直接与宿主免疫细胞表面的受体和抗体结合,引起机体强烈、持久的免疫反应,提供全面的保护作用,是理想的疫苗靶标。此外,病原体中存在许多帮助病原体逃逸宿主免疫系统识别和清除的分子,它们也能成为潜在的疫苗候选者。对病原体全基因组信息的解析是反向疫苗学技术“1.0 时代”的关键技术。随着蛋白质组学、结构生物学和免疫组学的快速发展,根据目标抗体的信息设计特异性较高的抗原成为可能,大大提高了疫苗研发效率[14],开启了反向疫苗学技术“2.0 时代”。
2.1 利用基因组进行抗原筛选
微生物基因组序列的全面解析,为反向疫苗学技术的发展奠定了基础。从基因组信息入手,先通过生物信息学分析算法找到病原体中能够编码成为潜在抗原的基因;再利用原核表达系统表达基因组分析找到的所有开放阅读框(Open Reading Frames,ORFs),最终纯化得到病原体实际表达的功能性蛋白;将纯化蛋白免疫动物后,应用多种免疫学检测技术评价其免疫原性和抗原性来进一步筛选。一般通过感染模型的攻毒实验评价候选抗原的保护能力,最终确定有效的保护性抗原;在完成临床前研究后,进一步将候选保护性抗原推进到临床试验研究。以上即反向疫苗学技术“1.0 时代”常用的技术路线,该方法已成功应用于B 群脑膜炎奈瑟菌疫苗研发中,已在多个国家和地区获得使用许可。
在反向疫苗学技术“1.0 时代”的研究中,最关键的环节是利用基因组学数据筛选和评估潜在抗原分子。随着新技术的不断涌现,生物信息学算法的迭代更新,研究人员可以从基因组数据中发掘更多的信息,包括抗原的表达和修饰信息、抗原的保守性分析以及病原体在流行病发展过程中的变异等。同时,随着蛋白质组学、免疫组学和结构生物学的发展,使得研究人员可以利用人源单克隆抗体信息鉴定保护性抗原和表位,并将抗原- 细胞表面受体、抗原- 中和抗体复合物的结构学表征用于指导抗原设计[13]。这样能够最大化利用基因组学数据,从多个维度筛选、评价和优化潜在抗原分子。至此,反向疫苗学技术进入“2.0 时代”,即进入多组学技术联用筛选抗原的时代。
2.2 基于生物信息学的基因组分析及抗原筛选
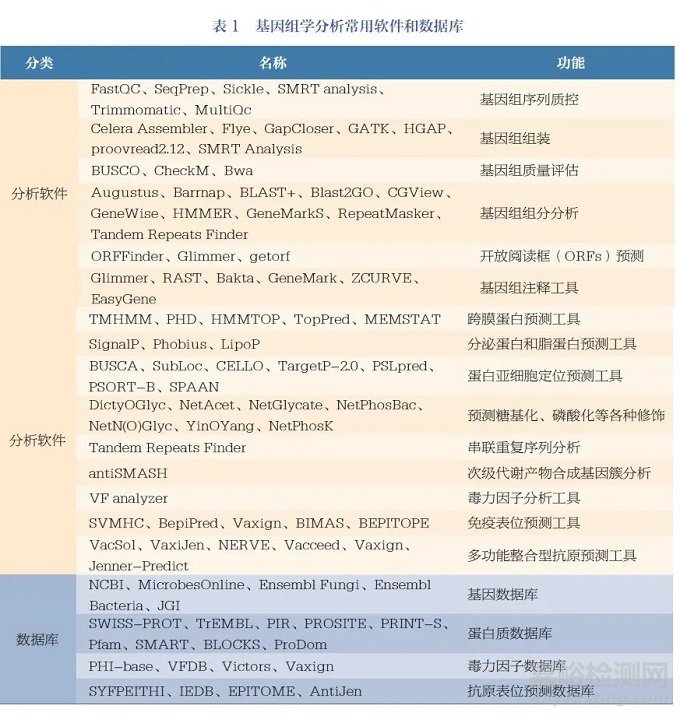
基因组分析的第一步是对目标病原体的基因组编码信息进行筛选和分析[1]。利用数据库和多种计算机程序筛选基因组序列,对DNA 片段和重叠信息进行整合, 进行基因组组分分析, 包括计算病原体基因组的编码容量、预测基因岛和非编码RNA等。第二步,预测所有的ORFs。通过BLASTX、BLASTN 和TBLASTX 等同源对比算法搜索数据库,鉴定出可以编码蛋白的DNA 片段[15-16]。将编码胞质功能或已知抗原的序列提出,对剩余编码区域进行下一步分析。第三步是通过同源性搜索和比对算法对编码蛋白进行亚细胞定位和功能注释。利用蛋白质数据库进行同源性检索——通过筛选目标蛋白所特有的典型特征元件、基序和指纹,如跨膜区结构域、信号肽、脂蛋白特征、外膜锚定元件和宿主细胞结合结构域,以及是否与已知的表面相关蛋白高度同源,筛选出病原体表面蛋白和毒素蛋白等[1]。常用的同源性搜索数据库包括BLAST、FASTA、MOTIFS 等[17],其他基因组学分析常用软件和数据库见表1。
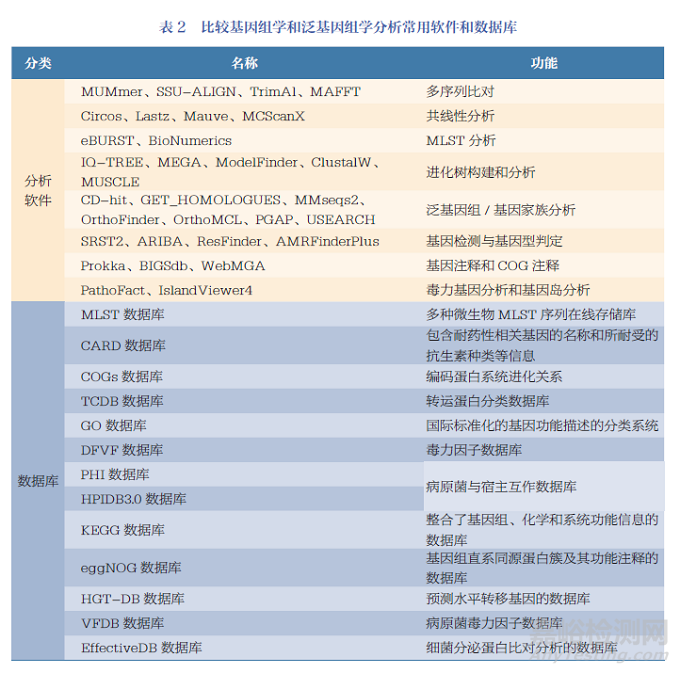
病原体为适应不同环境常会发生水平基因转移、相位变异和基因变异来逃逸宿主免疫系统的防御[18]。例如,在对同一病毒不同毒株的测序结果进行比对后,发现不同毒株的全基因组存在不同程度的差异[19]。因此,对于单一病原体的分析和筛选并不能很好地满足流行病学的发展需要。2005 年,泰特林(Tettelin)等人提出了微生物泛基因组(Pangenome)概念——泛基因组即某一物种全部基因的总称,而核心基因组(core genome)则是指在所有菌株中都存在的基因[20]。通过比较基因组学和泛基因组学,能够更加深入全面地认识种属间和种属内的抗原变化和分布情况[21],迅速鉴定出不同血清型和基因型中潜在的抗原分子[22],从而确定种属内保守性高的肽段和蛋白作为通用疫苗的候选分子[23]。此外,种间基因组学分析还能够指导研究人员筛选出与其他物种高度同源的基因,以避免不良的交叉反应,并剔除可能会引起宿主自身免疫病的候选分子[21]。
比较基因组学和泛基因组学分析的基本流程:高通量测序获得不同分离株的全基因组测序结果后,计算GC 含量,通过算法软件和数据库进行多角度分析[19, 24-33],其中包括MLST 分析、共线性分析、进化树构建和分析、毒力基因分析等,见表2。最终筛选出安全性高、高度保守且具有交叉保护能力的疫苗候选分子。
2.3 基于蛋白质组学和结构生物学的疫苗抗原预测和筛选
为适应动态多变的外界环境、逃逸宿主免疫系统的攻击,病原体的蛋白质表达谱在时空上会出现差异,也会发生多种翻译后修饰。这些复杂过程通常难以通过基因组数据分析进行准确地预测。而蛋白质组学分析能够弥补单一基因组学分析的误差,准确地研究病原体基因表达产物的组分和定位[34]。蛋白质组学在反向疫苗学技术中的应用主要包括病原体蛋白质组分分析、蛋白质定量分析、蛋白质修饰分析、抗原抗体相互作用和结构分析等方面。
在进行病原体蛋白质组分分析时,常通过双向凝胶电泳技术和质谱技术分离鉴定蛋白质组分。再配合蛋白质免疫印迹、酶解、多维色谱分离、蛋白质芯片等技术,可以从多维度分离和鉴定目标蛋白质群。而质谱技术的飞跃发展开启了大规模自动化的蛋白质鉴定时代, 先通过基质辅助激光解吸电离飞行时间质谱(MALDI-TOF-MS)、电喷雾电离质谱(ESI-MS)、表面增强激光解吸电离飞行时间质谱(SELDI-TOF-MS) 和傅里叶变换质谱法(FTMS)等技术鉴定多肽序列,再利用计算机算法在数据库中搜索相对应的氨基酸序列,最终鉴定目标蛋白[35]。以上方法已成熟运用于多种病原体疫苗的研发过程中,如用蛋白酶消化“刮掉”病原体表面的蛋白,再通过质谱方法鉴定这些蛋白组分,以筛选潜在的靶标蛋白[36]。
除了鉴定病原体的蛋白质组分,研究人员还开发出多种高通量测定蛋白质含量的方法,用以更准确地确定在致病等关键过程中起到重要作用的蛋白质。常用的蛋白质定量方法包括基于荧光染色技术的电泳定量法(F-2DDIGE)和质谱定量法。质谱定量法可以通过化学或代谢标签的标记[ 如同重元素标记的相对与绝对定量技术(iTRAQ)、多肽体外等重同位素标记的相对与绝对定量技术(TMT)、同位素代码标记技术(ICAT)和细胞培养条件下稳定同位素标记技术(SILAC)等方法] 定量比较不同样品中的蛋白质含量[37],也可以不进行标记,直接比较样品间的肽光谱计数或强度。定量蛋白组学可以通过SWATH 和msxDIA 快速定量分析数千种蛋白质,也可以通过选择性/ 多重反应监测(SRM/MRM)和平行反应监测(PRM)对复杂生物样品中的蛋白进行高度特异和灵敏的靶向定量分析[38]。
病原体蛋白质一般会发生多种修饰,包括磷酸化、糖基化和硫化等,这些修饰对于抗原抗体识别和免疫反应激活等过程是至关重要的。通过蛋白质组学对病原体蛋白的修饰情况进行研究,能为注释和研究蛋白质活性、定位和稳定性提供必要信息,而这些修饰信息无法通过基因组序列或mRNA 表达数据获得和预测。具体操作:酶解处理后的样品可以通过不同的吸附凝集柱(如金属螯合色谱柱)或特异性抗体进行富集,然后通过基质辅助激光解吸电离质谱(MALDI-MS)分析,计算谱峰偏移的距离,最终确定修饰的种类和数量[34]。
病原体的蛋白质组学分析还包括分析病原体蛋白和宿主受体之间的相互作用,通过荧光等特异性标记技术和蛋白质芯片技术,高通量捕获与宿主受体相互作用的病原体蛋白质和肽段,再联用基质辅助激光解吸(MALDI)技术读取和分析捕获的蛋白质数据。此外,还可以通过酵母双杂交技术[39] 和噬菌体展示技术[40] 等分析候选蛋白和靶标受体蛋白的结合力大小,优化疫苗候选蛋白的设计。
蛋白结构组学和计算生物学的发展,辅助免疫组学建立丰富的抗原表位肽库,成为反向疫苗学技术“2.0 时代”中最为突出的技术革新。通过蛋白质交联、小角散射和氢- 氘交换等技术辅助蛋白结构解析,利用X 射线、冷冻电镜和核磁共振等技术解析病原体蛋白结构和其与宿主受体相结合的复合物结构,帮助研究人员全面深入地了解免疫靶点[41]。通过构象变化和蛋白质结构数据库分析疫苗的候选蛋白,结合蛋白质化学、分子模型、人工智能和计算生物学,设计和优化疫苗候选蛋白, 最终得到免疫原性高、安全性高且保护效果好的蛋白分子。
2.4 基于免疫组学的抗原表位预测
分子免疫学和免疫信息学的快速发展,为研究人员提供了大量的病原体免疫表位方面的信息,推进反向疫苗学技术进入“2.0 时代”。抗原表位的预测在反向疫苗学技术“2.0 时代”是至关重要的,通过对病原体可能的免疫表位进行理论上的预测筛查,有助于缩小疫苗候选分子的范围,减少后续体内外试验所消耗的时间、人力以及物力。
抗原表位又称为抗原决定簇,是抗原物质分子表面或其他部位,具有一定组成和结构的特殊化学基团,能够与相应抗体或致敏淋巴细胞发生特异性结合。通过抗原表位预测工具设计出的理想疫苗应该能够刺激宿主产生B 细胞和T 细胞介导的免疫应答反应[42]。抗原表位预测方法主要包括两种:一种是基于蛋白质序列中氨基酸的理化特性(如亲/ 疏水性、柔性、表面可及性),另一种是基于人工智能算法进行预测。目前,研究人员一般会结合这两种预测方法进行抗原表位的预测、评估和筛选,最终找到具有高抗原特性和最佳理化特性的抗原表位, 再进入高通量蛋白表达和免疫检测环节。
2.4.1 B 细胞表位预测
虽然抗原- 抗体相互作用的研究有一百多年发展史,且早在20 世纪80 年代,B 细胞表位预测就受到了研究人员的重视,但基于生物信息学进行B 细胞表位预测的发展并不是很顺利。随着许多抗原- 抗体复合物和MHC-抗原肽-TCR 等复合物结构的解析,以及大量抗原肽表位资料的积累和计算机辅助设计的发展,基于人工智能算法的B 细胞表位预测得到了快速发展[43]。
目前, 预测B 细胞表位的数据库较多,见表3。这些数据库中所包含的B 细胞表位序列各不相同,各自的功能也具有一定的差异。例如,2017 年的一篇文献中指出BciPep 数据库包含555 个线性B 细胞表位序列,CED 数据库包含293 个不连续表位( 构象表位),Epitome 数据库是包含了所有已知抗原- 抗体复合物结构的数据[44]。因此,在进行B 细胞表位预测时, 仅依靠某一数据库进行预测可能是不充分的,需要结合不同的数据库,根据各个数据库得到的预测结果的分值,以确定最佳B 细胞表位候选分子。例如,阿米尔雷扎(Amirreza) 等利用IEDB、LBTOPE、ElliPro 和CBTOPE四种表位预测工具寻找新型冠状病毒候选抗原B 细胞表位,根据预测结果构建出了包含8 个表位的多表位抗原肽,并预测了其在用于血清学诊断产品开发等方面的巨大潜能[45]。
2.4.2 T 细胞表位预测
随着对细胞免疫应答机制的深入研究,许多研究人员将注意力集中于T 细胞表位预测上,希望能够开发出诱导产生细胞免疫应答的疫苗候选分子。分子免疫学和计算生物学的发展也提升了T 细胞表位预测的准确度。T 细胞免疫应答反应是由抗原呈递细胞表面的短肽(即抗原表位)刺激产生。这些表位的产生是通过抗原呈递细胞对抗原蛋白进行酶解加工产生抗原多肽,然后多肽结合到组织相容性复合体(major histocompatibility complex,MHC)分子的肽结合槽上,被转运到细胞表面进行展示,这些多肽与T 细胞受体结合,从而刺激T 细胞免疫应答反应[46]。
MHC 分子最主要的功能是参与抗原呈递,即通过其肽结合槽与抗原肽结合,将其呈现于细胞表面供T 细胞识别。MHC Ⅰ类分子介导的是内源性抗原的呈递过程, 其将内源性抗原片段呈递给细胞毒性T 淋巴细胞(cytotoxic T lymphocyte,CTL)。而MHC Ⅱ 类分子则呈递外源性抗原,其呈递外源性抗原多肽分子至Th 细胞(helper T cells)。此外,MHC Ⅰ 类分子几乎在所有细胞上都表达,这使得CTL 有机会检查机体内绝大多数细胞,以监视病原体。而MHC Ⅱ类分子则主要表达在B细胞、单核巨噬细胞、树突状细胞等抗原呈递细胞上,主要负责将危险警示于Th 细胞,协调免疫细胞间的相互作用,调控体液免疫和细胞免疫应答。
MHC Ⅰ类分子肽结合槽的两端呈闭合状态, 一般能够容纳8~10 个氨基酸残基组成的肽段。而MHC Ⅱ类分子肽结合槽的两端开放,多肽分子可以伸出沟外,这使其可容纳更长的肽配体分子, 一般可容纳13~18 个氨基酸组成的肽段。目前的预测工具对于MHC Ⅰ类分子结合肽的预测精确度接近95%,但是对于MHC Ⅱ类分子结合肽的预测精确度却相对较低。这是因为MHC Ⅱ类分子结合肽不像MHC Ⅰ类分子结合肽一样存在明确的基序,其锚点氨基酸的限制性也不如MHC Ⅰ类分子强,且由于MHC Ⅱ类分子可以利用的肽配体数据相对较少,使得对Th 细胞表位预测较之CTL 表位预测相对困难[47]。
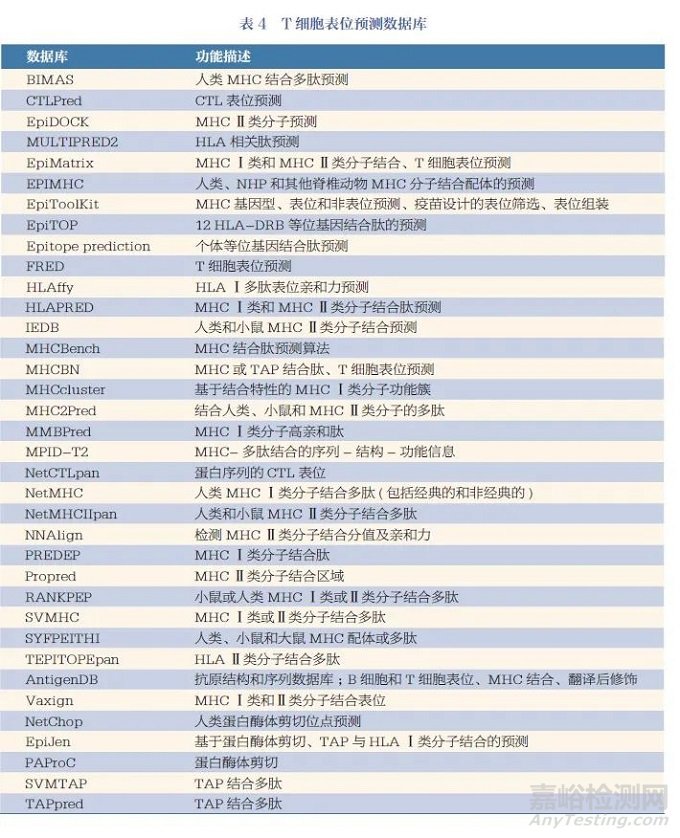
MHC Ⅰ类分子结合肽的预测方法包括两种:一种是基于抗原肽与MHC Ⅰ类分子结合特性的预测方法,另一种是针对抗原加工处理过程的预测方法。前者以MHC Ⅰ类分子晶体结构的解析为基础,后者以针对蛋白酶体裂解基序和抗原加工相关转运蛋白(transporter associated withantigen processing,TAP) 结合肽为基础。而MHC Ⅱ类分子的预测方法主要基于理化特性、抗原肽与MHC Ⅱ类分子结合特性。目前, 常用的T 细胞抗原表位预测数据库见表4。其中,NetChop、EpiJen 和PAProC常用于蛋白酶体裂解基序的预测,SVMTAP 和TAPpred 常用于TAP 结合多肽的预测。
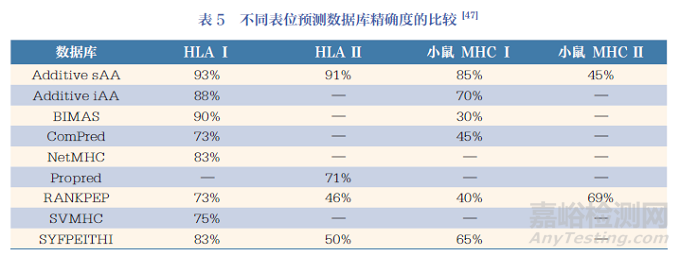
T 细胞表位预测时通常会选择多种数据库联合使用,以便更精准有效地预测出合适的T 细胞表位候选分子。马修(Matthew)等人对几个表位预测数据库进行比较, 结果显示这些数据库对Class Ⅰ HLA 的预测精确度高于Class Ⅱ HLA,对人的HLA 预测精确度高于小鼠的MHC,见表5。
另一项研究结果也发现,在对99 个已知表位用不同预测数据库进行检测时,EpiJen 能够鉴定出其中的61 个表位、SMM 鉴定出57 个、NetCTL 鉴定出49 个、WAPP 鉴定出33 个,且这几种预测方法得到的阳性预测值[ 阳性/(阳性+ 假阳性)] 都比较低,均低于25%[47]。
综上所述,在进行抗原表位预测时,无论是B 细胞表位还是T 细胞表位,均需要多种预测工具相结合,从理论上预测出可能包含B 细胞或T 细胞表位的候选分子。虽然任何预测软件均不能达到100% 准确性,或许存在预测分值较高但并不能真正刺激免疫应答反应的候选分子,但这些预测方法能大大降低寻找抗原表位的盲目性,降低试验成本[48]。
2.5 高通量筛选关键毒力因子
毒力因子是病原体产生的、导致宿主发病的一类分子,也是现用疫苗的主要成分之一。表面定位的毒力因子(如黏附蛋白)通过介导病原体的吸附,是开始感染宿主的过程。病原体产生的毒力因子一般具有很强的免疫原性,可能成为出色的靶点。通过多组学数据筛选和生物信息学计算预测得到的毒力蛋白,通常需要借助合适的动物和细胞模型进行验证。近年来,随着反向疫苗学技术的发展和成熟,研究人员主要通过两种思路来高通量筛选病原体的毒力基因:一种是构建基因突变株库筛选毒力基因,其中代表性方法为转座子随机突变技术和特征标签突变(STM)技术;另一种是通过毒力基因表达调控特点来进行体内诱导性筛选,代表性方法是差异荧光诱导技术(DFI)和体内诱导抗原技术(IVIAT)等。
通过转座子随机突变技术和STM 技术[49] 构建基因突变株库,根据突变株的毒力变化,高通量筛选与生物膜形成、黏附和致病相关的基因[50]。其中,STM 技术是一种更为常用的毒力基因筛选方法。该技术通过构建的突变株带有独特DNA 标签,感染动物模型后,相应感染位点或动物组织中分离不出的某个突变株就是减毒突变株。通过STM 技术反向筛选到的毒力基因可以研发成为减毒活疫苗和亚单位疫苗等。目前,已通过该方法找到多种病原细菌的毒力基因,如鼠伤寒沙门菌、B 群脑膜炎奈瑟菌和猪布鲁氏杆菌等[51]。
DFI 和IVIAT 等体内诱导性筛选方法是根据毒力基因特有的表达调控模式进行筛选,找到病原体在宿主体内表达而在体外不表达的功能基因,以此确定与病原体生存和致病关系密切的毒力因子。体内诱导性筛选思路主要包括三种:①通过体内表达技术(IVET)和DFI 等方法筛选具有体内诱导活性的启动子;②利用基因芯片比较体内外环境中基因转录水平的差异;③利用IVIAT等比较体内外环境中基因表达水平的差异。其中, IVET 的基本技术路线是将致病菌随机的DNA片段与无启动子的报告基因融合,构建融合基因文库,由质粒转入致病菌中进行整合,然后感染细胞或动物模型,激活体内诱导基因的表达。从感染细胞或动物体内分离致病菌,在平板上进行蓝白斑筛选找到在体外不表达的片段,最后进行测序和检索,确定体内诱导表达的启动子[52]。DFI是一种简化的体内表达技术[53-54],通过荧光标记和流式细胞仪筛选代替营养缺陷、抗生素或重组酶等筛选策略,通过改变荧光阈值调节筛选敏感度,以实现大规模半自动的高效筛选。IVET 和DFI方法曾被成功地用于福氏志贺菌、肺炎克雷伯菌和金黄色葡萄球菌等病原菌毒力因子的筛选。由于上述方法不适用于只对人致病且缺乏动物模型的病原菌,只能通过基因芯片技术和IVIAT 来进行研究,基因芯片技术通过比较病原体在宿主体内外的转录水平差异,以确定关键致病基因;IVIAT方法则通过体外培养的病原体吸附免疫血清,剔除患者血清中针对病原菌体外表达抗原的抗体,然后再用处理后的血清作为探针筛选病原体的基因组表达文库,获得体内诱导性表达基因[55]。这个方法在结核分枝杆菌和霍乱弧菌等的毒力因子筛选研究中取得了较大的进展。
2.6 其他多种新技术辅助筛选抗原
近年来,生物芯片技术得到迅猛发展,并应用到疫苗研制和药物开发过程中。生物芯片包括基因芯片和蛋白质芯片,将寡核苷酸、肽和抗体等生物高分子固定在凝胶和硅等固相介质上形成分子点阵,当待测样本中的生物分子与探针分子杂交或发生相互作用后,利用激光扫描共聚焦荧光显微镜或其他仪器检测和分析杂交信号[56]。基因芯片平台具有高通量、高灵敏度和全自动化等优势,可应用于病毒基因分型检测、点突变检测和核苷酸多态性研究中,助力多种病原菌的基因调控和毒力因子检测研究[57]。蛋白质芯片可以快速鉴定病原体在感染宿主过程中表达的蛋白[58]。生物芯片能够解决多种组学技术产生的繁冗数据,快速检测候选分子介导的宿主内细胞学事件和微生物致病进程,鉴定出效果优异的疫苗候选者[21]。在应对具有高度变异能力的病原体流行病学挑战中,生物芯片技术也凸显出了独特而重要的优势。
得益于芯片技术和测序技术的发展,转录组学能够通过RNA水平研究病原体和免疫细胞的基因表达情况。转录组测序技术(RNA-Seq)通量高、灵敏度高、分辨率高,且无需设计特异性探针,直接对任意对象和未知基因进行转录组分析,彻底转变了传统转录组学的研究思路[59-60]。转录组学和单细胞测序技术的飞速发展,使得单细胞转录组、空间转录组和单细胞免疫组库分析技术不断成熟,并广泛应用于疾病诊断和疫苗开发过程中。转录组学联合代谢组学和蛋白质组学技术,能够全方位、多层次、立体化地描述病原体感染宿主后发生的复杂生物学进程,大大加快新型疫苗的开发。
近年来,合成生物学脱颖而出,为新型疫苗的设计和生产开辟了新的策略,成为反向疫苗学技术研究中必不可少的重要工具。合成生物学能够应用于疫苗设计、开发和生产等各环节中,通过建立多种无细胞表达体系、定向改造工程生物和细胞模型来提高研发效率,同时降低生产成本。例如,研发人员通过合成生物学建立多种不具传染性的通用类病毒样颗粒(VLP)平台,结合结构设计和计算机建模等平台,合成、改造并优化候选疫苗分子的活性,提高候选蛋白的稳定性,开发出多种病毒结构蛋白和噬菌体作为靶向多种病原体的疫苗平台,为工程化疫苗的开发建立平台[61-64]。
疫苗开发过程中的重大进展往往是由发现新的候选抗原和优化免疫靶标的结构来驱动的,但具体过程非常复杂,需要联用多种技术手段共同推进。基于多组学的生物信息分析和免疫筛选结果确定候选分子,结合计算机建模、结构生物学和系统免疫学分析的结果,并将得到的信息应用于表位预测和靶标设计中,最终才能得到理想的疫苗候选分子。
3、 候选抗原有效性的分析与验证
3.1 候选抗原的高通量表达
基因组学分析后得到的部分候选基因并没有转录活性。为此, 研发人员优化出一种筛选具有转录活性的多聚酶链反应(transcriptionally activepolymerase chain reaction,TAP),通过连续PCR 技术扩增出目的基因,并在目的基因两侧添加启动子和终止子等表达调控元件。获得的TAP 片段能够模拟pcDNA3.1 表达质粒的活性,再通过转染在体内或体外系统中表达目的基因[65]。TAP 组分也可以用作DNA 疫苗免疫小鼠,筛选评价目的基因产物,还能够通过同源重组转入质粒进行体外功能探索。
通过多组学分析和多技术联用筛选得到的功能性基因,需要进行高通量的克隆表达,以鉴定候选分子的免疫原性。PCR 和体外自动化表达系统的发展为快速鉴定候选分子提供了方法。从目标基因序列设计入手,通过PCR扩增将目标基因克隆到大肠杆菌的表达载体上,融合多组氨酸标签或GST 等商业化标签,在原核细胞系统中进行高通量表达。通过简单的亲和柱色谱或离子亲和色谱,可快速纯化蛋白。对于表达在包涵体中的目标蛋白,可以通过变复性溶解和亲和色谱联用等方法,最终获得有活性的目标蛋白。目前,基因克隆和体外表达蛋白等各个阶段已经可以借助多种自动化仪器进入“一键式”高效表达阶段,极大提高了利用反向疫苗学技术开发新型疫苗的速度和效率。
利用原核细胞体外表达蛋白的方法虽然被广泛应用,但是该方法每一步都耗时耗力,存在一定的局限性。近年来,无细胞蛋白表达系统的兴起,给高通量表达蛋白的发展注入了“催化剂”。无细胞蛋白表达系统极大简化了操作步骤和下游实验流程[66],突破传统细胞内表达的极限,能够实现多模板和多系统的同时表达,还能够克服毒性蛋白、包涵体蛋白的表达难题, 完成多种标记修饰。此外, 无细胞蛋白表达系统还可以直接表达类病毒样颗粒(VLP),应用于病毒疫苗的开发[67]。
3.2 候选抗原的免疫原性检测
免疫原性是指能够刺激机体免疫系统产生免疫应答的特性,其能作用于B 淋巴细胞和T 淋巴细胞的抗原识别受体,促使细胞快速增殖、分化,从而产生免疫效应物质,如特异性抗体和致敏淋巴细胞。免疫原性检测是疫苗研发过程中作为评价疫苗有效性的一个重要参考指标,主要包括B 细胞免疫应答检测和T 细胞免疫应答检测。
3.2.1 B 细胞免疫应答检测
B 细胞是诱导体液免疫的细胞,主要通过分泌抗体产生体液免疫应答。它是由骨髓内多功能干细胞分化而来,需要在骨髓内发育及成熟,成为初始的成熟B细胞,之后随体液循环迁移到外周淋巴组织。若无相应抗原刺激,B 细胞会在几周后凋亡;若遇到相应抗原刺激,B 细胞将会活化、增殖进而分化成浆细胞产生抗体,而少数B 细胞会成为记忆细胞。B 细胞的活化根据抗原类型的不同可分为胸腺依赖性抗原和胸腺非依赖性抗原,顾名思义即需要Th 细胞直接接触B 细胞才能使其活化和不需要Th 细胞接触就能直接活化B 细胞。对于胸腺依赖性抗原,B 细胞在活化后,需要进入生发中心进一步增殖和分化。首先,活化的B 细胞迁移到滤泡中心进行周期性大量增殖和体细胞高频突变,形成细胞密集的生发中心暗区,体细胞高频突变发生在B 细胞识别抗原后的分裂增殖过程中,突变主要在B 细胞膜表面免疫球蛋白可变区的互补决定区,而这个互补决定区是决定抗体与抗原结合亲和力的关键。而后B 细胞迁移到生发中心明区,在滤泡Th 细胞和滤泡树突状细胞(follicular dendritic cell,FDC)的协助下进行亲和力成熟(即能够结合特异性抗原的具有高亲和力免疫球蛋白受体的B 细胞被选择存活)、抗体类别转化(即在细胞因子作用下,抗体可变区不变,恒定区发生改变,从而改变抗体类型,而不改变结合抗原的特异性)、分化成浆细胞和记忆B 细胞。浆细胞能够合成和分泌抗体,记忆B 细胞能够在相同抗原二次刺激后,迅速分化成浆细胞,从而产生大量抗体进行体液免疫[68]。
B 细胞免疫应答检测包括B细胞在生发中心成熟分化的过程,以及分泌抗体与抗原结合的反应检测。常用技术包括生发中心染色、活体组织成像技术、酶联免疫斑点试验(enzyme-linked immunospot assay,ELISPOT)、琼脂糖扩散法、酶联免疫吸附试验(enzyme-linked immunosorbent assay,ELISA)、免疫印迹等。
生发中心染色首先需要准备淋巴组织并进行特殊的处理(如液氮冷冻处理),之后通过激光显微切割技术制备切片,再用抗体对样品进行染色,常用的抗体包括CD21 和Ki67(CD21 为B淋巴细胞表面辅助受体,在B 细胞分化、增殖及抗原生成等方面起着重要作用;Ki67 是反应细胞增殖能力的一个重要指标),最后用荧光显微镜进行结果观察。活体全组织成像技术主要采用生物发光与荧光两种技术,生物发光是用荧光素酶报告基因标记细胞或DNA,利用其产生的蛋白酶与相应底物发生生化反应产生生物体内的探针光信号;而荧光成像则是采用荧光报告基团或荧光染料进行标记,利用荧光蛋白或染料产生的荧光形成体内的荧光光源。该技术应用非常灵敏的光学检测仪器能够直接监测活体生物体内的细胞活动和基因表达,跟踪目标的移动和变化,如通过对淋巴结或B 细胞标记可以追踪生发中心内淋巴结或B 细胞的迁移情况。ELISPOT 即将细胞置于包被有特异性抗体板子中,在有刺激的条件下,细胞分泌的细胞因子或免疫球蛋白会被包被抗体捕获,移除细胞后,被捕获的细胞因子或免疫球蛋白可进一步使用生物素标记的第二抗体进行标识,形成“抗体- 抗原- 抗体”复合物,其后再与酶标记亲和素作用,并加入底物使其呈色,形成斑点即说明细胞产生了细胞因子或免疫球蛋白。该技术是检测分泌细胞的一种重要手段。
抗原抗体反应是抗原与相应抗体在体内外发生的特异性结合反应。检测抗原抗体反应的经典方法是琼脂糖扩散法,其原理是可溶性抗原与相应抗体在半固体琼脂凝胶内扩散,二者相遇,在比例合适处形成白色沉淀线,一条沉淀线即代表一种抗原与抗体的沉淀物,因而此法还可以用于鉴别多种抗原或抗体成分。另一种常用的方法是免疫酶技术,就是将抗原抗体反应特异性与酶的高效催化作用有机地结合起来。其是把具有催化活性的酶类和抗体(抗原)结合起来成为酶标记物,将这种酶标记物与待测的抗原(抗体)结合,通过酶的活性降解底物呈现出颜色从而对抗原(抗体)进行定性或定量分析。目前, 应用最多的免疫酶技术是ELISA,其是使抗原或抗体吸附于固相载体,使随后进行的抗原抗体反应均在载体表面进行,从而简化了分离步骤,提高了灵敏度,既可检测抗原,也可检测抗体。实验方法包括间接法、夹心法及竞争法。常用的酶包括辣根过氧化物酶(HRP)和碱性磷酸酶(AP),相应的底物分别是邻苯二胺(OPD)和对硝基苯磷酸盐(pNPP),前者呈色反应为棕黄色,后者为蓝色,可用于目测定性,也可用于酶标仪测定光密度(OD)值以反映抗原或抗体的含量。还有一种方法是免疫血清的特异性免疫印迹检测,即将抗原蛋白或细菌提取物进行蛋白质凝胶电泳及转膜,之后免疫血清作为第一抗体孵育,HRP 标记的抗体作为第二抗体孵育,最后用发光成像仪进行显影即可观察到抗原抗体是否特异性结合。
3.2.2 T 细胞免疫应答检测
T 细胞免疫应答通常检测CTL 活性、T 细胞增殖和细胞因子。CTL 活性检测最初使用的是铬释放测定法,该方法由于使用放射性的51Cr 不利于安全操作及废物处理,且需特殊测定仪器,因此研究人员一直试图寻找其他的替代方法。目前,CTL 活性检测方法主要包括荧光测定法、报告基因转染法和比色测定法。
荧光测定法又可分为荧光扫描测定法、流式细胞分析法和树突状细胞清除法[69]。其中, 荧光扫描测定法包括alamarBlue一步荧光测定法和Calcein-AM 荧光扫描测定法[70-71]。流式细胞分析法包括PE-mAb/FITC-annexin V 荧光标记法、DIOC18 (3)/ 碘化丙啶(PI) 荧光标记法、PKH-26/CFSE 荧光标记法[72-74]。这些方法灵敏度高、重复性好,适用于大批量样品的测定。报告基因转染法则需要构建报告基因稳定转染细胞系,该方法灵敏度高,不同报告基因转染的细胞系可同时测定CTL 杀伤活性且结果互不干扰。但该方法也有不足之处,如制备细胞系耗时费力,且报告基因在一些靶细胞中有时会出现难以转染或表达的情况[75]。除了上述方法,还有比色测定法,如MTT 法或MTS 法和乳酸脱氢酶(LDH)释放法[76-77]。另外也有一些其他的CTL 活性测定方法,如“ 鸡尾酒” 混合刺激法、ELISPOT 等[78-80]。
T 细胞增殖检测的方法主要包括氚标淋巴细胞增殖测定法(3H-TdR 掺入法)、MTT 法、MTS 法和XTT 比色法。由于氚标记法需要使用放射性同位素,且购买和使用都有相关要求,也需要专业的操作人员。因此,目前使用较为普遍的是MTT、MTS和XTT 这三种方法。MTT 法又称MTT 比色法,其检测原理为活细胞线粒体中的琥珀酸脱氢酶能使外源性MTT 还原为水不溶性的蓝紫色结晶甲瓒并沉积在细胞中,而死细胞无此功能,且结晶形成的量与细胞数成正比[81]。MTS和XTT 原理与MTT 一样,且已经有商品化的MTS 细胞增殖试剂盒,这使得T 细胞增殖检测更方便。
细胞因子检测方法主要包括实时定量PCR(real time PCR,RT-PCR) 技术、ELISPOT 和流式分析法。RT-PCR 主要用于检测细胞因子的mRNA 水平,此方法需要相应的引物和荧光报告分子,且检测的是细胞因子基因而非细胞因子本身,该方法具有操作简单、特异性强、灵敏度高等特点[82]。ELISPOT 和流式分析法是基于免疫学检测的两种方法,原理是细胞因子与相应的特异性抗体结合,通过荧光或酶等标记技术将信号放大,从而定性或定量检测细胞因子水平,这两种方法具有操作简单快捷、特异性高、重复性好等特点[83-84]。
3.3 候选抗原的免疫效果评价
确定抗原有效保护力最直接的方法是进行动物试验。因为有些具有免疫原性的抗原可能不具有保护作用,或者虽然具有保护作用但会出现一些不良反应。因此,疫苗效果评价是决定疫苗成功与否的最后关键步骤。免疫效果评价的方法主要包括动物流行病学方法、血清学方法和动物攻毒保护试验。
动物流行病学方法即对免疫动物和非免疫动物的生长表现、生化指标、病死率等临床指标进行调查统计,通过统计学分析比较,评价疫苗的免疫保护效果。生长表现包括体重变化、体温变化等。生化指标包括注射部位是否出现充血、肿胀、硬结等现象,血常规、尿常规、心电图等指标是否异常。
血清学方法包括中和抗体滴度检测以及抗体中和试验。抗体滴度是用来衡量某种抗体识别特定抗原表位所需要的最低浓度(即最大稀释度),一般表示为能产生阳性结果的最大稀释度,常用ELISA 检测抗体滴度。通过比较免疫前后抗体滴度升高的幅度及持续时间来评价疫苗的免疫保护效果。抗体中和试验是将病毒或毒素与中和抗体进行混合孵育,使二者发生反应,再将二者的混合物接种到敏感的细胞中,测定残存的病毒或毒素感染力的一种方法。抗体中和试验具有较高的特异性,利用同一病毒(病原菌)的不同型毒株(菌株)或不同型标准血清,即可测得相应血清或毒株(菌株)的型,因此该方法常被研究人员用于鉴定病毒(或病原菌)的种或型。
动物攻毒保护试验即在免疫一段时间后,用病原菌或病毒感染动物模型,攻毒后通过观察动物的临床表现、检测攻毒后中和抗体的变化、病原菌或病毒载量的差异以及组织病理学检测等指标从而评价疫苗的保护效果。动物的临床表现包括观察是否出现明显临床症状、是否出现体重急剧变化、是否出现发病或死亡的情况等。攻毒后的抗体水平检测和上述血清学方法相同。病原菌或病毒载量的检测一般采用RTPCR法,即提取待检测样品(血液或组织等)中的RNA,用特异性的引物和探针对RNA 样品进行RT-PCR 扩增,最后通过比较免疫组和非免疫组中的病原菌或病毒载量之间的差异评价疫苗的保护效果。组织病理学检测主要采用组织病理切片方法。该方法是将部分有病变的组织或脏器经过各种化学品和包埋剂的处理,使之固定硬化,在切片机上切成薄片,最后染以各种颜色,在显微镜下观察病理变化。常用的染色方法是苏木素- 伊红(hematoxylin-eosin)染色法,简称HE 染色法。苏木素是一种碱性染料,可使组织中的嗜碱性物质染成蓝色,如细胞核中的染色质等。伊红是一种酸性染料,可使组织中的嗜酸性物质染成红色,是胞质、胶原纤维、肌纤维、嗜酸性颗粒等常用的染料。HE 染色法对任何固定液固定的组织和应用各种包埋法的切片均可使用。常规苏木素染色中的对比染色是用伊红,近年来在英国、美国的一些实验室则采用荧光桃红(phloxine),此外也有用桔黄G(orange G)、比布里希猩红(biebrich scarlet)、波尔多红(bordeaux red)等作为对比染色。
4、 结语
反向疫苗学技术的飞速发展为复杂的病原体开发新型有效的疫苗、攻克流行性疾病开辟了新思路。反向疫苗学技术具有大规模、高通量、自动化等特点,充分利用基因组学、蛋白质组学、免疫组学以及生物信息学等手段筛选潜在的疫苗候选分子,并鉴定出具有保护性的抗原,在防治新型突发传染病方面具有一定的优势。对于部分病原体,利用传统疫苗方法研究依旧存在瓶颈的,建议可以积极尝试利用反向疫苗学技术进行新型疫苗的研发。