您当前的位置:检测资讯 > 科研开发
嘉峪检测网 2025-02-28 08:10
生命科学领域的研究者常常需要回答一个核心问题:DNA上的调控元件如何像"开关"一样控制基因活动?
面对这个挑战,科学家们发明了ChIP-seq、ATAC-seq等表观遗传学工具。但传统技术如同用渔网捕捞特定鱼类——效率低、背景噪音高。
直到2019年,Steven Henikoff团队在《Nature Communications》发表的CUT&Tag技术,犹如为表观研究装上了"分子显微镜",让染色质调控研究进入高精度时代。

图片来源:https://images.app.goo.gl/gGUgbbHkr8NE4okD7
一、CUT&Tag技术原理:当抗体成为"分子导航"
如果把染色质比作一本折叠的立体书,那么CUT&Tag就是精准定位书中特定段落的技术。其核心原理可拆解为四个精密设计的步骤:
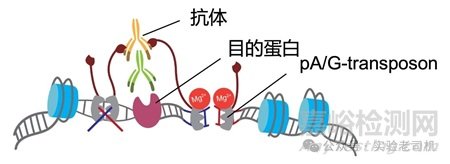
1. 活细胞定位(Live-cell targeting)
实验从完整细胞开始,加入目标蛋白(如H3K27me3修饰组蛋白)的特异性抗体。这些抗体如同带有GPS的分子探测器,在37℃生理条件下精准锚定靶点,避免传统ChIP-seq中甲醛交联导致的空间结构破坏。
2. 蛋白A-Tn5转座酶融合体的"分子剪刀"
蛋白A能特异性结合抗体的Fc段,携带经过工程化改造的Tn5转座酶。这种酶经过特殊设计,平时处于休眠状态,只有在结合镁离子后才激活DNA剪切功能。这种"触发式"设计极大降低了背景噪音。
3. 靶向DNA片段化(Tagmentation)
加入Mg²⁺激活Tn5后,转座酶在抗体结合位点附近同时完成DNA双链切割和测序接头连接。这一过程产生的DNA片段大小约300-500bp,完美覆盖目标蛋白的结合区域。
4. 片段释放与测序
加入SDS和EDTA终止反应,通过蛋白酶K消化核膜后,靶向切割的DNA片段即可释放。由于非特异性切割极少,文库构建效率较传统ChIP-seq提升10倍以上。
图片来源:https://images.app.goo.gl/HCZPrRk2ihok4ZDk6
二、技术优势:三项突破性创新
1. 信噪比革命
CUT&Tag的背景信号仅为ChIP-seq的1/10。这得益于:
无需超声破碎(减少随机断裂)
抗体引导的靶向切割
实验全程在完整细胞/细胞核内完成
2. 微量样本适应性
传统ChIP-seq需要10⁶级细胞量,而CUT&Tag仅需500-50,000个细胞即可获得高质量数据。2021年改进的单细胞CUT&Tag技术甚至能在单个细胞水平解析组蛋白修饰图谱。
3. 分辨率跃升
结合二代测序,CUT&Tag可精确定位到±50bp范围内的结合位点。在端粒等重复序列区域,其分辨率优势尤为明显。
三、应用场景:解码生命调控的"暗物质"
1. 干细胞命运抉择的分子密码
北京大学邓宏魁团队利用CUT&Tag绘制了重编程过程中H3K4me3的动态变化,发现特定增强子的提前激活预示细胞命运决定,为再生医学提供新靶点。
2. 癌症表观异质性解析
MD Anderson癌症中心的研究显示,乳腺癌中CTCF结合位点的表观异质性可通过CUT&Tag精准捕获,这种异质性与化疗耐药性显著相关。
3. 三维基因组学新维度
结合Hi-C技术,CUT&Tag可同时获得特定蛋白结合与染色质空间互作信息。例如,2023年《Cell》的一项研究揭示了Cohesin蛋白在染色质环形成中的动态招募过程。
图片来源:https://images.app.goo.gl/PFNqRyase8kHfpZo7
四、挑战与未来:通往临床应用的最后一公里
尽管优势显著,CUT&Tag仍面临三大挑战:
1. 标准化难题:不同实验室的抗体批次差异可能导致数据偏差,亟需建立类似ENCODE项目的统一质控标准。
2. 多组学整合瓶颈:如何与ATAC-seq、RNA-seq数据实现时空同步分析仍是方法论难点。
3. 临床转化障碍:尽管已实现石蜡切片样本检测,但检测灵敏度仍需提升以适应微量循环肿瘤DNA分析。
未来发展方向呈现三大趋势:
单细胞多组学整合:如10x Genomics推出的Single Cell Multiome ATAC + CUT&Tag方案。
空间表观组学:结合空间转录组技术,在组织原位解析表观调控网络。
动态过程捕获:开发时间分辨型CUT&Tag,追踪发育/疾病进程中的实时表观变化。
五、技术对比:CUT&Tag vs 传统ChIP-seq
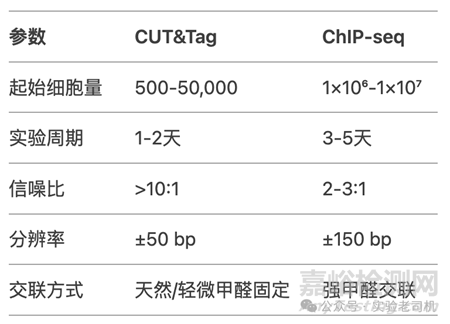
总结:表观遗传学的"纳米手术刀"
正如冷冻电镜革命性地改变了结构生物学,CUT&Tag技术正在重塑表观遗传学研究范式。它不仅是一个实验方法,更代表了一种研究理念的转变——从"大规模筛选"到"精准定位",从"群体平均"到"单细胞解析"。
随着空间组学、人工智能等技术的融合,CUT&Tag有望在疾病诊断、药物开发等领域展现更大潜力。当科学家们手握这把"分子手术刀",染色质调控的"暗宇宙"正逐渐显现出清晰的星图。
参考文献:
Kaya-Okur HS et al. (2019)
CUT&Tag for efficient epigenomic profiling of small samples and single cells.
Nat Commun 10:1930. doi:10.1038/s41467-019-09982-5
Meers MP et al. (2019)
Improved CUT&RUN chromatin profiling tools.
eLife 8:e46314 . doi:10.7554/eLife.46314
Wang Q et al. (2021)
Low-input CUT&Tag reveals dynamic histone modifications during early embryogenesis.
Cell Res 31:701–704. doi:10.1038/s41422-021-00487-9
Bartosovic M et al. (2021)
Single-cell CUT&Tag profiles histone modifications and transcription factors in complex tissues.
Nat Biotechnol 39:825–835. doi:10.1038/s41587-021-00869-9
Deng H et al. (2022)
Dynamic H3K4me3 mapping reveals epigenetic priming during iPSC reprogramming.
Cell Stem Cell 29:135–150. doi:10.1016/j.stem.2021.12.002
Rao SSP et al. (2023)
Cohesin-mediated loop anchors confine CTCF recruitment in 3D genome folding.
Cell 186:1236–1252. doi:10.1016/j.cell.2023.02.010
10x Genomics (2023)
Single Cell Multiome ATAC + CUT&Tag Solution.

来源:实验老司机煲仔饭


