一、 背景
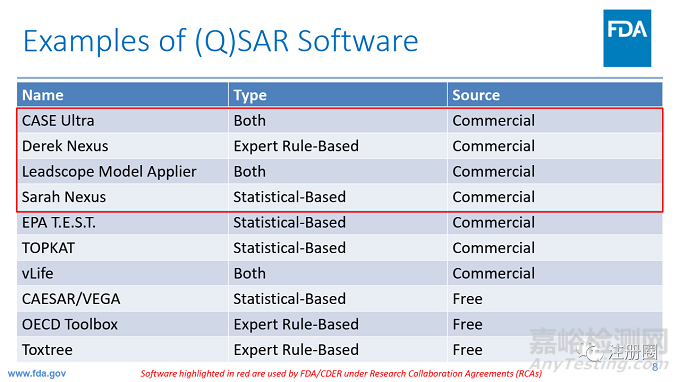
1)利用(Q)SAR软件进行化合物的致突变性评估,无需进行复杂的毒性试验,已越来越广泛地应用于药物的基因毒性杂质评估中。商业化的软件目前市面上有好几家,如最常用的CASE Ultra,Derek&Sarah,Leadscope等。但上述软件的授权费用较贵,且委外评估费用也较高,基于目前杂质需要全评估的趋势,对企业来说是一笔不小的开支。其实也有一些免费的QSAR软件,其不仅预测准确率较高,且也被欧美法规机构所认可,不失为基因毒性杂质初步评估的一个选择。
2)根据ICH-M7章节6中的描述:“危害性评估首先通过对数据库和文献的检索获得的杂质致癌性和细菌致突变数据对实际和潜在杂质进行初步分析,根据表 1 将其归为 1 类, 2 类或 5 类。如果无法获得这样的分类数据,则应进行预测细菌致突变性的构-效关系( SAR)评估。根据评估结果将杂质归为 3 类、 4 类或 5 类。”
指南该段话对企业而言,即应这样进行危害性评估:先文献和数据库检索杂质的相关致癌性和Ames数据,然后根据结果分类为1,2,5类;如果无法找到相关数据,应进行QSAR评估,根据评估结果分类为3类,4类和5类。
在FDA2021年3月进行了DMF培训中,Drug Master File and Drug Substance Workshop – FDA CDER SBIA Events
在day2: ICH M7(R1) – Chemistry and Manufacturing Control (CMC) Perspective on Hazard Assessment – Barbara O. Scott 中明确指出:通过目视判断化合物警示结构分类是不符合ICH-M7规定的。
3)在近期的各国监管评审过程中,已收到多次缺陷,需要对所有使用的物料和杂质进行致突变性评估,即使它没有警示结果或者推测肯定是无致突性的。
Deficiency-1:QSAR or genotoxicity information for all matters
Recent trend of document examination by PMDA, QSAR for all matters is required even if they do not have alert structure.
Deficiency-2:Provide the genotoxicity data (experimental data or (Q)SAR report) of all the reagents, starting materials, intermediates and related substances involved in the manufacturing of API(MFDS)
二、 评估方法
1)采用毒性数据库,对常见的商业化物料进行毒性数据检索,重点进行基因毒性检索及致癌性数据检索。上述检索可通过常用的一些免费数据库来进行,也可通过免费的QSAR TOOLBOX 软件来进行。
最常用的3个网站如下:
Lhasa Carcinogenicity Database:可查询物质的致癌性数据及TD50值,查询后需对结果进行判断,判断数据是否充分且符合ICH S1的要求。
NTP database:可查询物质的致癌性数据及体外体内基因毒性数据,Ames实验需判断是否满足OECD的指南要求,同时关注有无代谢活化情况下的结果差异。
ECHA database: 可在该物质的toxicological information中查看Genetic toxicity 和Carcinogenicity
QSAR Toolbox的数据查询功能
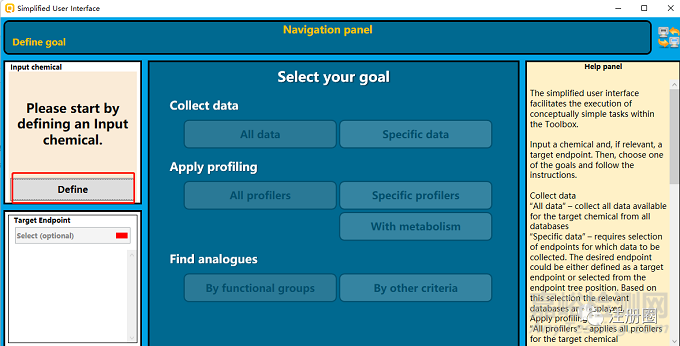
a.选用简易用户模式,进入结果,点击Define输入目标化合物
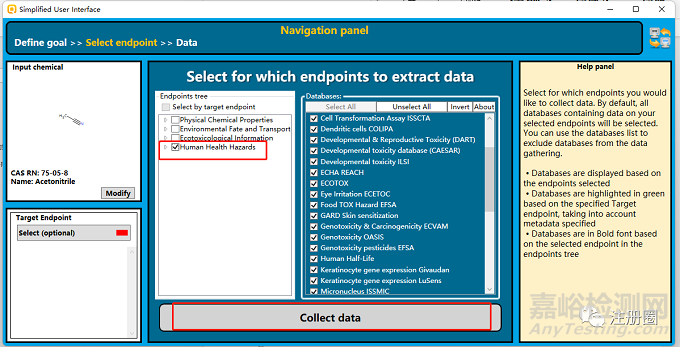
b.点击Specific data, 勾选Human health hazards,然后点击Collect data
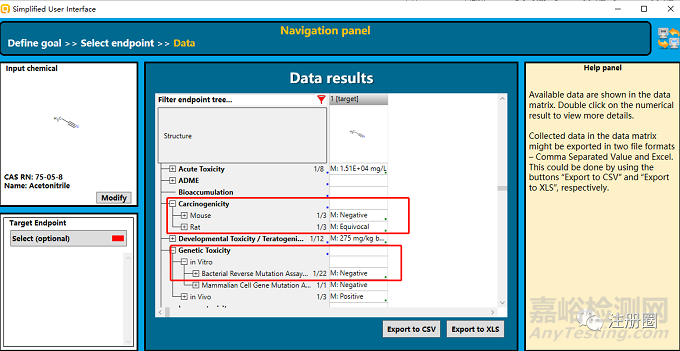
c.在data results 界面可查询对应类别的实验性毒性数据,重点关注遗传毒性和致癌性,可点击相应的数据查看数据的source 和reference。
2)采用FDA认可的免费QSAR评估软件进行评估。FDA认可的QSAR软件如下:
统计学规则:CAESAR/VEGA
https://www.vegahub.eu/portfolio-item/vega-qsar/
专家规则:Toxtree, OECD Toolbox,
Toxtree: https://toxtree.sourceforge.net/
OECD Toolbox: https://qsartoolbox.org/download/
对于免费QSAR软件评估接受性的担忧,可参考FDA 2021年3月进行了DMF培训中说明:
CDER已经对这些免费的QSAR有了前期的知识储备,因此可以不用再递交经过验证的QMRF,可直接进行结果说明即可。
Drug Master File and Drug Substance Workshop – FDA CDER SBIA Events
Application of (Q)SAR and Expert Knowledge for ICH M7 Impurity Classification– Naomi L. Kruhlak
三、 免费QSAR软件具体使用方法:
根据ICH-M7章节6中的描述:“计算机毒理学应采用(定量)构-效关系((Q)SAR)方法学进行毒性评估,目的是预测细菌致突变试验(参考文献 6)的结果。应采用两种互补的(Q)SAR 预测方法。一种方法应基于专家知识规则,另一种方法应基于统计学。(Q)SAR 模型采用的这些预测方法学应遵循经济合作与发展组织(OECD)制订的一般的验证原则。如果经两种互补的(Q)SAR 方法(专家知识规则和统计学)预测均没有警示结构,则足以得出该杂质没有致突变性担忧的结论,不建议做进一步的检测(归为表 1 中的 5 类)。”
因此我们需要采用不同原理的两种QSAR软件来对杂质进行评估,具体的策略如下:
-----采用Toxtree或QSAR Toolbox 进行专家规则的评估
-----采用VEGA进行统计学规则的评估
-----若两个评估结果都为阴性,可直接判断为5类杂质;
如其中一个为阳性,建议对该杂质采用商业化评估软件(如CASE Ultra,derek&sarah,leadscope)进行评估,以商业化评估软件出具的结果为准。
3.1 Toxtree的使用方法
软件最新版本:Toxtree 3.1.0
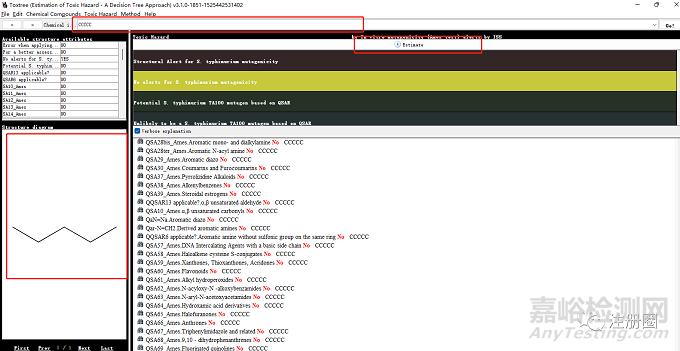
决策树选择:点击最上栏中的method,点击select a decision tree,选择:In vitro mutagenicity (Ames test) alerts by ISS,然后点下方确定按键。
化合物键入:在Chemdraw中将化合物的结构转化为SMILES, 复制后输入搜索框,核对左下角的结构是否与目标结构一致,然后点击Estimate 运行。
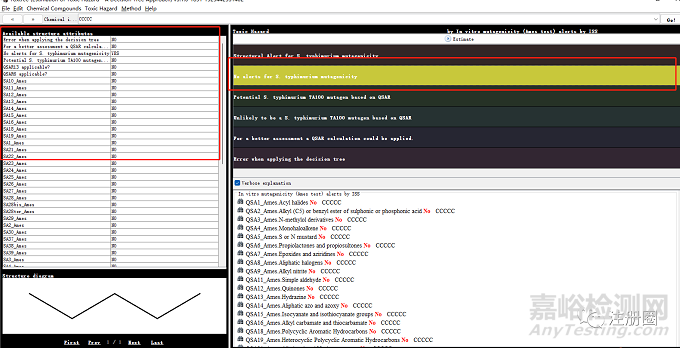
观察左边窗口的结果及中间的色条显示,若结论为:No alerts for S.typhimurium mutagenicity,则表示为阴性。
3.2 VEGA的使用方法
软件最新版本:
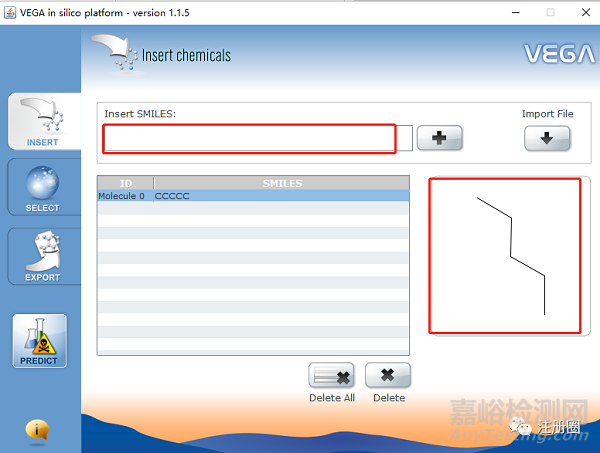
化合物键入:在Chemdraw中将化合物的结构转化为SMILES, 复制后输入搜索框,点击+号,核对右边显示的结构是否与目标杂质一致。
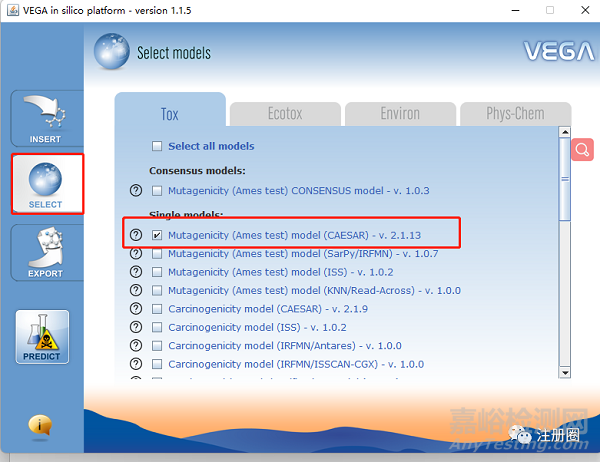
模型选择:点击左边SELECT,然后选择Mutagenicity(Ames test)model(CAESAR)-V.1.0.3
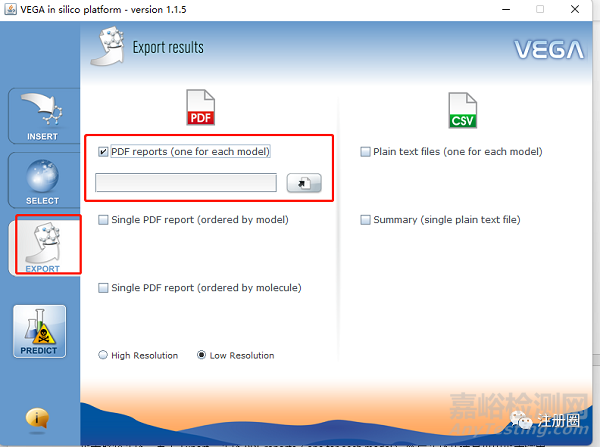
报告路径选择:点击Export,选择PDF reports(one for each model),然后选择在计算机中报告输出的路径。
预测:点击PREDICT进行预测,在设定的路径下找到生成的预测报告,根据报告中小球的颜色及下方的评论,判断预测的结果。绿色表示阴性,红色代表阳性。
3.3 QSAR Toolbox的使用方法
由于QSAR Toolbox与Toxtree为同一规则,且使用的决策树均为In vitro mutagenicity (Ames test) alerts by ISS,两者预测结果是一样的。考虑到Toxtree为java形式运行,使用更方便,建议直接用Toxtree即可。
杂质评估结果在CTD资料中的书写格式:
参照如下格式在CTD资料中提交杂质的hazard assessment summay,在comments 中加入experimental data source ,具体使用的QSAR软件和相关的控制策略简述。
参考文献:
[1] ICH. Assessment and control of DNA reactive (mutagenic) impurities in pharmaceuticals to limit potential carcinogenic risk M7(R2). International Council for Harmonisation of Technical Requirements for Pharmaceuticals for Human Use (ICH)
[2] Honma M, Kitazawa A, Cayley A, et al. Improvement of quantitative structure–activity relationship (QSAR) tools for predicting Ames mutagenicity: Outcomes of the Ames/QSAR International Challenge Project. Mutagenesis. 2019; 34(1): 3– 16.
[3] Cassano, A., Raitano, G., Mombelli, E., Fernández, A., Cester, J., Roncaglioni, A., Benfenati, E., 2014. Evaluation of QSAR models for the prediction of Ames genotoxicity: a retrospective exercise on the chemical substances registered under the EU REACH regulation. J. Environ. Sci. Health C 32, 273–298.