您当前的位置:检测资讯 > 实验管理
嘉峪检测网 2025-02-20 08:39
一、单细胞测序的概念及特点
单细胞测序技术是指在单个细胞的水平上,对基因组、转录组等组学信息进行高通量测序分析的一种技术。但是,单细胞测序的总量并不仅仅是几个细胞那么简单,根据样本类型和测序需求,可以在单细胞水平下对成千上万的细胞进行测序。既然总量这么大,为什么还要进行单个细胞分辨率的测序呢?为什么不一次性把组织内的所有细胞都检测完然后进行分析呢?
这是因为尽管多细胞生物是由一个细胞不断增殖分化而来的,但随着这一过程的深入,细胞与细胞之间会产生异质性,甚至我们已经认定的同一类型细胞当中,也会有或大或小的差异,这些差异决定了细胞真正的类型和功能。
传统测序技术是在组织水平(至少是在多细胞水平)上进行的,得到的数据也是这些细胞的平均值,因此没法得知细胞的异质性。而单细胞测序可以能够揭示每个细胞的基因表达状态,从而得到异质性信息。
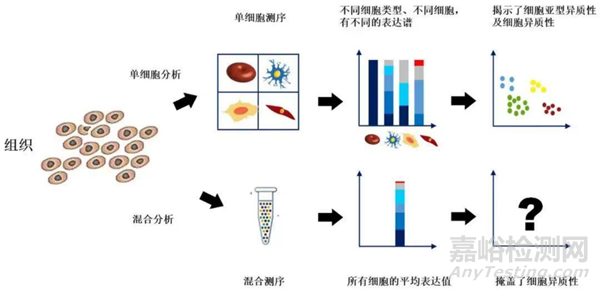
(图片来源:10x Genomics)
二、单细胞检测策略
目前,单细胞的检测主要有两种策略:
1.通过流式细胞术(主要用于细胞样品)或者激光显微切割(LCM)技术(主要用于组织切片)分离单个细胞,构建测序文库,最后进行测序。
这种方法通量低,且随着待测细胞数目的增多,测序成本也会大幅提升。但临床上的样本细胞数量动辄10^9个以上,想要以几十个细胞的信息代表临床样本,显然是不合适的。
2.基于barcode测序。将单细胞悬液和带有barcode的珠子包裹在一个油滴之中,并对其进行逆转录,这样每个单细胞的cDNA文库就带上了特异性的barcode。最后将所有单细胞的cDNA文库混在一起测序,同时通过程序识别barcode,区分出单细胞。
这种方法可以通过一次建库,测到成百上千个细胞的信息。
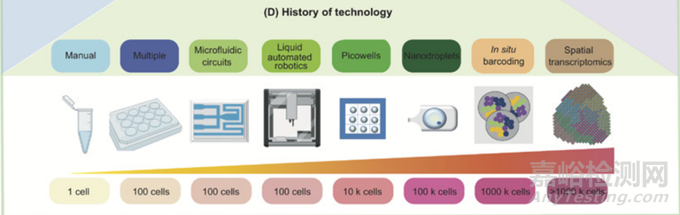
(Single‐cell RNA sequencing technologies and applications: A brief overview, Clinical and translational medicine 10.1002/ctm2.694)
三、单细胞的分离制备(以肿瘤组织为例)
1.制备单细胞悬液:
(1)一般采用脱颈处死,解剖过程务必快速准确,尽量保留细胞的活性;
(2)使用预冷过的能够维持细胞活性的液体(如生理盐水/PBS/RPMI-1640等)冲洗组织;
(3)剪碎组织:根据后续测序的需要(优先保证细胞数量还是质量)选择合适体积的组织进行切碎,过程中注意预冷;
(4)常规消化离心:这一步在处理不同组织、不同细胞时的注意事项较多,建议多查阅文献;
(5)红细胞裂解:像心脏、肝脏、脾脏等红细胞含量丰富的组织难免需要裂解红细胞。根据上面切碎的组织大小加入裂红液裂红,结束后用PBS终止。如果裂解不干净,可以重复一次,但不能超过两次。离心重悬后即可获得单细胞悬液。
2.样本细胞筛选:
单细胞筛选的方法包括:激光捕获显微分离(laser capture microdissection,LCM)、显微微管操作法(Micropipetting)、流式细胞分选(fluorescence activated cell sorting,FACS)、有限稀释法(Limiting dilution)、微流控技术(microfluidics)等。方法各有优劣,可根据需要灵活选择。
(1)有限稀释法
有限稀释法是一种通过连续稀释细胞群来获得单细跑的方法,此方法操作简便,且不需要特殊的设备,但是依赖于梯度稀释计算,不是直观的单个细胞分离,容易出现错误。这种方法可以说是最简单、也是最不准确的单细胞分离的方法,很少用于复杂样品的单细胞分离。
(2)显微微管操作法
当细胞样品的复杂度较低时,可以采用显微操作技术根据细胞的形态待征来分选细跑。目前主要用于分离一些难以培并的微生物细跑,但是由于其通量低、人力成本高、容易造成细胞的机械损伤等缺点,难以广泛应用。
(3)激光捕获显微分离
当目的细胞在细跑群中呈分散分布,旦占有的比例很小时,一般采用激光捕获显微分离(LCM) 技术,直接从组织中分离单细跑。这种技术在临床医学中应用比较广泛。但由于操作过程中切割精度有限,容易掺杂相邻细跑的原生质,以及目的细胞容易丢失核染色体片段。因此在转录组学分析中,一般不采用这种技术。
(4)微流控技术
与其他方法比较, 微流控平台相结合单细胞测序技术在降低单细胞测序的噪声以及基因组扩展更加均匀方面具有优势, 显示出了良好的应用前景。在物理学上,流体惯性可以在微米级尺度上忽略,微流控技术正是利用这一原理,人为控制(细胞)流体的流动,来实现单细胞的分离。但是尽管微流控芯片技术因样品用量小而可以有效避免污染,但是由于其成本较高,故实际应用并不广泛。
(5)流式细胞分选
流式细胞分选技术是目前最流行的单细胞分离技术。流式系统按照细胞特征 (大小、颜色、核酸、生物化学活性等) 迅速将单个细胞分选到几百个孔板中。但是, 这种方法也有固有的缺陷: 一是需要大量的细胞悬浮液, 会降低低丰度细胞的获得率;二是分选速率比较快, 容易造成细胞损伤。
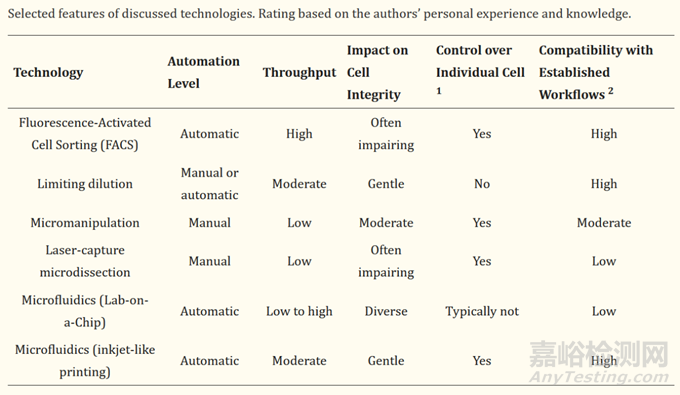 (Technologies for Single-Cell Isolation. International journal of molecular sciences, 10.3390/ijms160816897)
(Technologies for Single-Cell Isolation. International journal of molecular sciences, 10.3390/ijms160816897)
四、遗传物质的提取扩增
单细胞测序起始样本中核酸含量极低,典型的哺乳动物细胞包含大约10 pg总RNA, 其中mRNA仅有大约0.1~0.5 pg, 而目前深度测序技术需要纳克级别的DNA, 因此文库构建过程需要将起始的核酸扩增数十万倍。
目前主要的单细胞测序扩增方法有:单细胞全基因组扩增和单细胞全转录组扩增。
全基因组扩增(WGA)是将从单细胞中提取出的DNA进行扩增以获取大量DNA样品的技术。如今已研发出多种扩增方法,例如基于 PCR法的简并寡核苷酸引物PCR(DOP-PCR)、基于等温扩增法的多重置换扩增(MDA)、基于PCR法和等温扩增法的多次退火环状循环扩增技术(MALBAC)。
利用PCR技术进行全基因组的扩增,扩增的覆盖度比较高,但容易出现错误和覆盖度不均的现象,因此该技术目前已经在逐步淘汰。
多重置换扩增技术能在常温下扩增,避免了高温下DNA降解对扩增产物质量的影响及GC含量不同引发的优势扩增。但是此方法的扩增均匀性不高。多重置换扩增技术可用于单核苷酸多态性以及突变相关的研究。
多次退火环状循环扩增技术是目前最先进的全基因组扩增技术,基因组覆盖率和扩增的均匀性都显著高于其他技术,因此常用于那些样本稀少、用常规方法无法进行基因组或转录组分析,或者样本高度异质,细胞之间存在重要差异的样本。

单细胞全转录组扩增(Whole transcriptome amplification, WTA)方法需要将单细胞内的mRNA提取后,通过反转录成cDNA,然后对cDNA进行扩增。一般常采用PCR指数扩增、体外转录线性扩增和Phi29聚合酶扩增三种方法。
五、高通量测序
下面这张图是单细胞测序数据分析内容的概述。该图显示了单细胞测序数据实际分析中的分析内容的总结,其可以分为四个分析模块,分别是(A)数据预处理模块,(B)通用分析模块,(C)探索性分析模块和(D)可选分析模块。
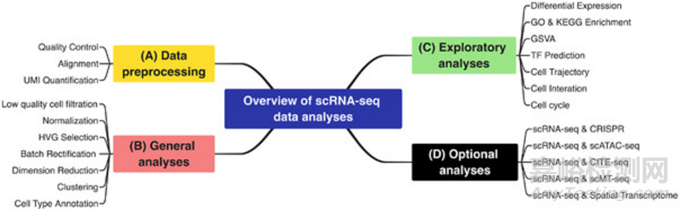
对单细胞测序数据的分析是另一个重要部分,到了这一部分就进入了生物信息学的研究领域。一般多花一些钱测序公司会负责产出配套的数据分析,根据难度不同价格也不同。比如你根据前期文献调研,已经确定了自己想要的数据分析图片类型和图片数量,那么价格会相对低一些。但如果你没有明确的分析方向,需要让测序公司的数据分析人员同步跟进你的课题进展,价格自然会变高。
另外一种选择,自学数据分析。目前网上有着非常多数据分析的教学视频和配套代码,并且也有越来越多的公共单细胞数据可以获取,对于非常多缺少经费的课题组而言,无疑提供了非常好的数据资源。在上游的数据预处理和初步统计分析方面,推荐推荐大家去学习python,在后续部分,R语言是进行组学数据的统计分析及可视化最推荐学习的工具。对于医学背景或者生物学背景的同学而言,数据分析和基础实验/临床实验相结合的干湿结合方法是发文的主流,因此我们并不需要在数据分析领域作根本性的创新,只需要正确的理解和运用即可。因此只要跟着正确的方法,就可以快速掌握。

来源:实验老司机


